
Dědičná informace uložená v DNA (genom) není u většiny organizmů tvořena pouze geny. Většinu genomu tvoří záhadná „temná hmota“ představovaná opakujícími se úseky DNA neboli repeticemi. Hlavními repeticemi jsou tzv. transpozony (často označované jako skákající geny) a tandemově uspořádané repetice neboli satelity. Repetice nejsou v genomu uspořádány rovnoměrně, ale často se shlukují v oblasti centromer nebo na koncích chromozomů. Bylo otázkou, zda toto hromadění repetic v určitých oblastech genomu je důsledek jejich náhodného včleňování a následného zachování procesem selekce, anebo jsou naopak repetice pouze do určitých míst chromozomů cíleně včleňovány, například do centromer.
V rámci projektu, podpořeného Grantovou agenturou ČR (GA ČR), tým docenta Kejnovského ukázal, že transpozony nejsou včleňovány do genomu náhodně, ale jsou přednostně včleňovány do jiných transpozonů (tzv. „nesting“), a to dokonce do sobě podobných transpozonů. Tato místa přednostního včleňování dokázali v rámci výzkumu nalézt. Příkladem takového místa jsou palindromatické sekvence DNA, místa, která se čtou v obou směrech stejně¹ (příkladem palindromu je například věta „jelenovi pivo nelej“ nebo „kobyla má malý bok“). Přednostní včleňování do již existujících transpozonů dává z evolučního hlediska smysl, neboť právě v těchto místech transpozony genomu nejméně škodí.
V rámci projektu řešitel a jeho tým vytvořili program „TE Greedy Nester“ určený pro hledání LTR retrotranspozonů, tedy transpozonů, které se šíří v genomech duplikativně prostřednictvím molekul RNA.² Jejich program si, na rozdíl od konkurenčních programů, poradí i s obřími genomy zamořenými transpozony, jako je genom kukuřice, tvořený až z 80 % transpozony.
U transpozonů je důležité také správné určení jejich evolučního stáří. Vědecký tým zjistil, že klasická metoda určování věku LTR retrotranspozonů založená na divergenci dlouhých koncových repetic (LTR, jsou při včlenění shodné) není přesná v důsledku genové konverze. Proto navrhl vylepšenou metodu určování stáří těchto transpozonů, která proces genové konverze zohledňuje.³
Řešitelský tým dokázal také zjistit, že transpozony velmi často obsahují motivy, které ochotně tvoří čtyřvláknovou DNA, pro jejíž hledání vytvořili nový software.⁴ DNA se totiž vyskytuje nejen v ikonické podobě známé dvoušroubovice, ale také jako struktura třívláknová (triplex), nebo dokonce čtyřvláknová (kvadruplex). Bylo prokázáno, že kvadruplex má regulační roli a funguje jako přepínač různých molekulárních procesů v buňce. Ve spolupráci se skupinou prof. Kateryny Makove z PennState University v USA vědci zjistili, že kvadruplexové motivy zpomalují, nebo dokonce zastavují prodlužování DNA při sekvenování DNA moderní metodou PacBio.⁵ Navíc odhalili, že v místech kvadruplexových motivů se nachází nejvíce mutací DNA, jak ve zdravých, tak zejména v nádorových buňkách. Rovněž zjistili, že kvadruplexy inhibují skákání Ty1 elementů u kvasinky.⁶
Transpozony patří mezi nejdynamičtější složky genomu. Při řešení projektu se tedy řešitelé zamýšleli nad otázkou, proč jsou některé složky evolučně dynamičtější než jiné. Odpovědí je jejich publikace⁷ prezentující hypotézu, že příčinou vyšší evoluční dynamiky některých genomových elementů je jejich větší náchylnost k migraci či pohybu po buňce i mezi buňkami. Největší mobilitu vykazují právě transpozony, tandemové repetice a promiskuitní DNA (tj. DNA migrující mezi buněčnými organelami chloroplasty/mitochondriemi a jádrem).
Všechny uvedené a během výzkumu zkoumané složky genomu jsou pak významnými hráči fungování genomu a buňky. Poskytují genomům variabilitu, která je předpokladem evoluce a adaptace a kterou člověk také odpradávna využívá při šlechtění kulturních plodin.
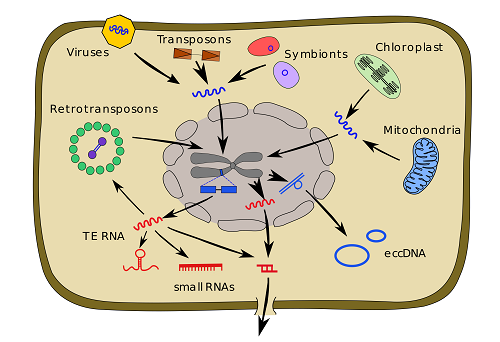 Pohyb evolučně nejdynamičtějších složek genomu po buňce. EccDNA – extrachromozomální cirkulární DNA, TE RNA – přepis transpozonu. DNA je modře, RNA je červeně (autor obrázku Pavel Jedlička).
Pohyb evolučně nejdynamičtějších složek genomu po buňce. EccDNA – extrachromozomální cirkulární DNA, TE RNA – přepis transpozonu. DNA je modře, RNA je červeně (autor obrázku Pavel Jedlička).
Výstupem projektu je také několik odborných článků:
¹ Jedlička et al 2019, Mobile DNA
² Lexa et al 2020, Bioinformatics
³ Jedlička et al 2020, Front Plant Sci
⁴ Labudová et al 2020, Bioinformatics
⁵ Guiblet et al 2018, Genome Research
⁶ Tokan et al 2018, BMC Genomics; Tokan et al 2021, Biology
⁷ Kejnovsky and Jedlička 2022, BioEssays
Autor článku: doc. RNDr. Eduard Kejnovský, CSc.